Diagnóstico de la enfermedad de Wilson y sus fenotipos usando inteligencia artificial
DOI:
https://doi.org/10.54502/msuceva.v3n1a5Palabras clave:
Ciclo de Krebs, ciclo de la urea, cobre, hígado, metabolismo intermediario, mitocondria, red neuronal artificialResumen
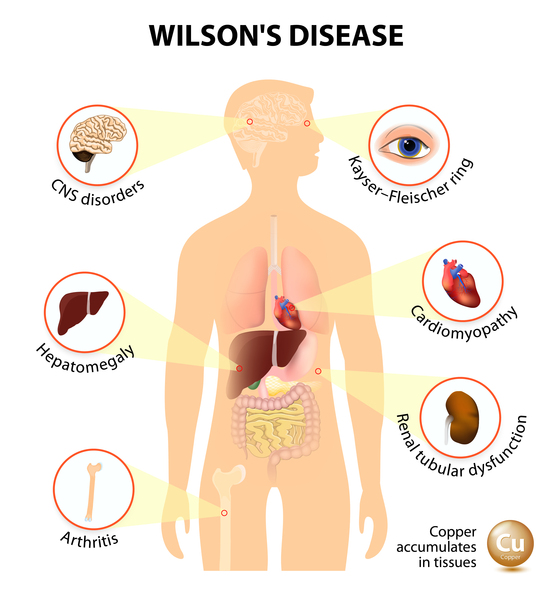
La EW es causada por las variantes de ATP7B que alteran el eflujo de cobre y provocan una acumulación excesiva de cobre, principalmente en el hígado y el cerebro. El diagnóstico de la EW se ve dificultado por su evolución clínica variable, su aparición, su morbilidad y el tipo de variante ATP7B. Actualmente se diagnostica mediante una combinación de síntomas/signos clínicos, parámetros aberrantes del metabolismo del cobre (por ejemplo, niveles séricos bajos de ceruloplasmina y concentraciones elevadas de cobre urinario y hepático) y cuando están disponibles, a través de pruebas genéticas de mutaciones ATP7B. Dado que el diagnóstico y el tratamiento precoces son clave para obtener resultados favorables, es fundamental identificar a los sujetos antes de la aparición de manifestaciones clínicas manifiestamente perjudiciales. Con este fin, tratamos de mejorar el diagnóstico de la EW mediante algoritmos de redes neuronales artificiales (parte de la inteligencia artificial) integrando los parámetros clínicos y moleculares disponibles. Sorprendentemente, el diagnóstico de la EW se basó en los niveles plasmáticos de glutamato, asparagina, taurina y el cociente de Fischer. Dado que estos aminoácidos están relacionados con los ciclos urea-Krebs, nuestro estudio no sólo subraya el papel central de las mitocondrias hepáticas en la patología de la EW, sino también que la mayoría de los pacientes con EW presentan una disfunción hepática subyacente. Nuestro estudio aporta pruebas novedosas de que la inteligencia artificial utilizada para el análisis integrado de la EW puede dar lugar a un diagnóstico más precoz y a tratamientos mecánicamente relevantes para los pacientes con EW.
Descargas
Métricas
Citas
Harris, E.D. Cellular copper transport and metabolism. Annu. Rev. Nutr. 2000, 20, 291–310.
https://doi.org/10.1146/annurev.nutr.20.1.291 DOI: https://doi.org/10.1146/annurev.nutr.20.1.291
Jayakanthan, S.; Braiterman, L.T.; Hasan, N.M.; Unger, V.M.; Lutsenko, S. Human copper transporter ATP7B (Wilson disease protein) forms stable dimers in vitro and in cells. J. Biol. Chem. 2017, 292, 18760–18774.
https://doi.org/10.1074/jbc.M117.807263 DOI: https://doi.org/10.1074/jbc.M117.807263
Gromadzka, G.; Schmidt, H.H.-J.; Genschel, J.; Bochow, B.; Rodo, M.; Tarnacka, B.; Litwin, T.; Chabik, G.; Czlonkowska, A. Frameshift and nonsense mutations in the gene for ATPase7B are associated with severe impairment of copper metabolism and with an early clinical manifestation of Wilson’s disease. Clin. Genet. 2005, 68, 524–532.
https://doi.org/10.1111/j.1399-0004.2005.00528.x DOI: https://doi.org/10.1111/j.1399-0004.2005.00528.x
Panagiotakaki, E.; Tzetis, M.; Manolaki, N.; Loudianos, G.; Papatheodorou, A.; Manesis, E.; Nousia-Arvanitakis, S.; Kanavakis, E. Genotype-phenotype correlations for a wide spectrum of mutations in the Wilson disease gene (ATP7B). Am. J. Med. Genet. 2004, 131A, 168–173. https://doi.org/10.1002/ajmg.a.30345 DOI: https://doi.org/10.1002/ajmg.a.30345
Czlonkowska, A.; Litwin, T.; Dzieżyc, K.; Karliński, M.; Bring, J.; Bjartmar, C. Characteristics of a newly diagnosed Polish cohort of patients with neurological manifestations of Wilson disease evaluated with the Unified Wilson’s Disease Rating Scale. BMC Neurol. 2018, 18, 1–6. https://doi.org/10.1186/s12883-018-1039-y DOI: https://doi.org/10.1186/s12883-018-1039-y
Czlonkowska, A.; Litwin, T.; Dusek, P.; Ferenci, P.; Lutsenko, S.; Medici, V.; Rybakowski, J.K.; Weiss, K.H.; Schilsky, M.L. Wilson disease. Nat. Rev. Dis. Prim. 2018, 4, 1–20.
https://doi.org/10.1038/s41572-018-0018-3 DOI: https://doi.org/10.1038/s41572-018-0018-3
European Association for the Study of the Liver. EASL Clinical Practice Guidelines: Wilson’s disease. J. Hepatol. 2012, 56, 671–685. https://doi.org/10.1016/j.jhep.2011.11.007 DOI: https://doi.org/10.1016/j.jhep.2011.11.007
Ferenci, P.; Czlonkowska, A.; Merle, U.; Ferenc, S.; Gromadzka, G.; Yurdaydin, C.; Vogel, W.; Bruha, R.; Schmidt, H.T.; Stremmel, W. Late-Onset Wilson’s Disease. Gastroenterology 2007, 132, 1294–1298.
https://doi.org/10.1053/j.gastro.2007.02.057 DOI: https://doi.org/10.1053/j.gastro.2007.02.057
Lorincz, M.T. Neurologic Wilson’s disease. Ann. New York Acad. Sci. 2009, 1184, 173–187. https://doi.org/10.1111/j.1749-6632.2009.05109.x DOI: https://doi.org/10.1111/j.1749-6632.2009.05109.x
Steindl, P.; Ferenci, P.; Dienes, H.P.; Grimm, G.; Pabinger, I.; Madl, C.; Dobersberger, T.M.; Herneth, A.; Dragosics, B.; Meryn, S.; et al. Wilson’s disease in patients presenting with liver disease: A diagnostic challenge. Gastroenterology 1997, 113, 212–218. https://doi.org/10.1016/S0016-5085(97)70097-0 DOI: https://doi.org/10.1016/S0016-5085(97)70097-0
Czlonkowska, A.; Tarnacka, B.; Litwin, T.; Gajda, J.; Rodo, M. Wilson’s disease—cause of mortality in 164 patients during 1992–2003 observation period. J. Neurol. 2005, 252, 698–703.
https://doi.org/10.1007/s00415-005-0720-4 DOI: https://doi.org/10.1007/s00415-005-0720-4
Chen, L.; Liu, R.; Liu, Z.-P.; Li, M.; Aihara, K. Detecting early-warning signals for sudden deterioration of complex diseases by dynamical network biomarkers. Sci. Rep. 2012, 2, 1–8.
https://doi.org/10.1038/srep00342 DOI: https://doi.org/10.1038/srep00342
Scheffer, M.; Bascompte, J.; Brock, W.A.; Brovkin, V.; Carpenter, S.R.; Dakos, V.; Held, H.; van Nes, E.; Rietkerk, M.; Sugihara, G. Early-warning signals for critical transitions. Nature 2009, 461, 53–59. https://doi.org/10.1038/nature08227 DOI: https://doi.org/10.1038/nature08227
Jiang, L.; Sui, D.; Qiao, K.; Dong, H.-M.; Chen, L.; Han, Y. Impaired functional criticality of human brain during Alzheimer’s disease progression. Sci. Rep. 2018, 8, 1–11.
https://doi.org/10.1038/s41598-018-19674-7 DOI: https://doi.org/10.1038/s41598-018-19674-7
Liu, X.; Chang, X.; Liu, R.; Kazuyuki, A.; Chen, L.; Aihara, K. Quantifying critical states of complex diseases using single-sample dynamic network biomarkers. PLoS Comput. Biol. 2017, 13, e1005633.
https://doi.org/10.1371/journal.pcbi.1005633 DOI: https://doi.org/10.1371/journal.pcbi.1005633
Liu, X.; Liu, R.; Zhao, X.-M.; Chen, L.; Liu, X.; Liu, R.; Zhao, X.-M.; Chen, L. Detecting early-warning signals of type 1 diabetes and its leading biomolecular networks by dynamical network biomarkers. BMC Med. Genom. 2013, 6, S8.
https://doi.org/10.1186/1755-8794-6-S2-S8 DOI: https://doi.org/10.1186/1755-8794-6-S2-S8
Lu, L.; Jiang, Z.; Dai, Y.; Chen, L. Low-grade dysplastic nodules revealed as the tipping point during multistep hepatocarcinogenesis by dynamic network biomarkers. Genes 2017, 8, 268. https://doi.org/10.3390/genes8100268 DOI: https://doi.org/10.3390/genes8100268
Teschendorff, A.E.; Liu, X.; Caren, H.; Pollard, S.M.; Beck, S.; Widschwendter, M.; Chen, L. The dynamics of DNA methylation covariation patterns in carcinogenesis. PLoS Comput. Biol. 2014, 10, e1003709. https://doi.org/10.1371/journal.pcbi.1003709 DOI: https://doi.org/10.1371/journal.pcbi.1003709
Yang, B.; Li, M.; Tang, W.; Liu, W.; Zhang, S.; Chen, L.; Xia, J. Dynamic network biomarker indicates pulmonary metastasis at the tipping point of hepatocellular carcinoma. Nat. Commun. 2018, 9, 1–14. https://doi.org/10.1038/s41467-018-03024-2 DOI: https://doi.org/10.1038/s41467-018-03024-2
Czlonkowska, A.; Gajda, J.; Rodo, M. Effects of long-term treatment in Wilson’s disease with D-penicillamine and zinc sulphate. J. Neurol. 1996, 243, 269–273. https://doi.org/10.1007/BF00868525 DOI: https://doi.org/10.1007/BF00868525
Gromadzka, G.; Chabik, G.; Mendel, T.; Wierzchowska, A.; Rudnicka, M.; Czlonkowska, A. Middle-aged heterozygous carriers of Wilson’s disease do not present with significant phenotypic deviations related to copper metabolism. J. Genet. 2010, 89, 463–467.
https://doi.org/10.1007/s12041-010-0065-3 DOI: https://doi.org/10.1007/s12041-010-0065-3
Medici, V.; Sarode, G.V.; Napoli, E.; Song, G.; Shibata, N.M.; Guimarães, A.O.; Mordaunt, C.E.; Kieffer, D.A.; Mazi, T.A.; Czlonkowska, A.; et al. mtDNA depletion-like syndrome in Wilson disease. Liver Int. 2020, 40. https://doi.org/10.1111/liv.14646 DOI: https://doi.org/10.1111/liv.14646
Mazi, T.A.; Sarode, G.V.; Czlonkowska, A.; Litwin, T.; Kim, K.; Shibata, N.M.; Medici, V. Dysregulated choline, methionine, and aromatic amino acid metabolism in patients with Wilson Disease: Exploratory metabolomic profiling and implications for hepatic and neurologic phenotypes. Int. J. Mol. Sci. 2019, 20, 5937.
https://doi.org/10.3390/ijms20235937 DOI: https://doi.org/10.3390/ijms20235937
Sarode, G.V.; Kim, K.; Kieffer, D.A.; Shibata, N.M.; Litwin, T.; Czlonkowska, A.; Medici, V. Metabolomics profiles of patients with Wilson disease reveal a distinct metabolic signature. Metabolomics 2019, 15, 1–12. https://doi.org/10.1007/s11306-019-1505-6 DOI: https://doi.org/10.1007/s11306-019-1505-6
Perlich, C.; Provost, F.; Simonoff, J. Tree induction vs. logistic regression: A learning-curve analysis. J. Mach. Learn. Res. 2004, 4, 211–255. https://www.jmlr.org/papers/volume4/perlich03a/perlich03a.pdf
Quinlan, J.R. Induction of decision trees. In Machine Learning; Kluwer Academic Publishers: Boston, MA, USA, 1986; pp. 81–106. DOI: https://doi.org/10.1007/BF00116251
https://link.springer.com/content/pdf/10.1007/BF00116251.pdf
Shannon, C.E. The mathematical theory of communication. 1963. MD Comput. 1997, 14, 306–317. PMID: 9230594.
Giulivi, C.; Zhang, Y.-F.; Omanska-Klusek, A.; Ross-Inta, C.; Wong, S.; Hertz-Picciotto, I.; Tassone, F.; Pessah, I.N. Mitochondrial
dysfunction in autism. JAMA 2010, 304, 2389–2396. https://doi.org/10.1001/jama.2010.1706 DOI: https://doi.org/10.1001/jama.2010.1706
Liang, L.-P.; Patel, M. Plasma cysteine/cystine redox couple disruption in animal models of temporal lobe epilepsy. Redox Biol. 2016, 9, 45–49. https://doi.org/10.1016/j.redox.2016.05.004 DOI: https://doi.org/10.1016/j.redox.2016.05.004
Zhou, Z.; Jia, R.-X.; Zhang, G.; Wan, Y.; Zhang, Y.; Fan, Y.; Wang, Z.; Huang, P.; Wang, F. Using cysteine/cystine to overcome oxidative stress in goat oocytes and embryos cultured in vitro. Mol. Med. Rep. 2016, 14, 1219–1226.
https://doi.org/10.3892/mmr.2016.5395 DOI: https://doi.org/10.3892/mmr.2016.5395
Fischer, J.E.; Rosen, H.M.; Ebeid, A.M.; James, J.H.; Keane, J.M.; Soeters, P.B. The effect of normalization of plasma amino acids on hepatic encephalopathy in man. Surgery 1976, 80, 77–91. PMID: 818729.
Medici, V.; Kieffer, D.A.; Shibata, N.M.; Chima, H.; Kim, K.; Canovas, A.; Medrano, J.F.; Islas-Trejo, A.D.; Kharbanda, K.K.; Olson, K.; et al. Wilson Disease: Epigenetic effects of choline supplementation on phenotype and clinical course in a mouse model. Epigenetics 2016, 11, 804–818.
https://doi.org/10.1080/15592294.2016.1231289 DOI: https://doi.org/10.1080/15592294.2016.1231289
Chong, J.; Wishart, D.S.; Xia, J. Using MetaboAnalyst 4.0 for comprehensive and integrative metabolomics data and analysis. Curr. Protoc. Bioinform. 2019, 68, e86. https://doi.org/10.1002/cpbi.86 DOI: https://doi.org/10.1002/cpbi.86
Nazer, H.; Ede, R.J.; Mowat, A.P.; Williams, R. Wilson’s disease: Clinical presentation and use of prognostic index. Gut 1986, 27, 1377–1381. http://dx.doi.org/10.1136/gut.27.11.1377 DOI: https://doi.org/10.1136/gut.27.11.1377
Francioso, A.; Conrado, A.B.; Mosca, L.; Fontana, M. Chemistry and Biochemistry of Sulfur Natural Compounds: Key Intermediates of Metabolism and Redox Biology. Oxid. Med. Cell. Longev. 2020. https://doi.org/10.1155/2020/8294158 DOI: https://doi.org/10.1155/2020/8294158
Piussan, C.; Mathieu, M. Teratogenic risk during treatment of Wilson disease. J. Genet. Hum. 1985, 33, 357–362. PMID: 4056754.
Lheureux, P.; Penaloza, A.; Gris, M. Pyridoxine in clinical toxicology: A review. Eur. J. Emerg. Med. 2005, 12, 78–85. DOI: https://doi.org/10.1097/00063110-200504000-00007
Ortiz, J.F.; Cox, M.; Tambo, W.; Eskander, N.; Wirth, M.; Valdez, M.; Niño, M. Neurological Manifestations of Wilson’s Disease: Pathophysiology and Localization of Each Component. Cureus 2020, 12.
https://doi.org/10.7759/cureus.11509 DOI: https://doi.org/10.7759/cureus.11509
Packman, S. Wilson’s Disease. In Encyclopedia of the Neurological Sciences; Aminoff, M.J., Daroff, R.B., Eds.; Academic Press: New York, NY, USA, 2003; pp. 759–763. DOI: https://doi.org/10.1016/B0-12-226870-9/00026-5
Metsalu, T.; Vilo, J. ClustVis: A web tool for visualizing clustering of multivariate data using Principal Component Analysis and heatmap. Nucleic Acids Res. 2015, 43, W566–W570.
https://doi.org/10.1093/nar/gkv468 DOI: https://doi.org/10.1093/nar/gkv468
Lough, J.; Wiglesworth, F.W. Wilson disease. Comparative ultrastructure in a sibship of nine. Arch. Pathol. Lab. Med. 1976, 100, 653–659. PMID: 1036679.
Suzuki, K.; Ogura, Y. Biological regulation of copper and selective removal of copper: Therapy for Wilson disease and its molecular mechanism. Yakugaku Zasshi 2000, 120, 899–908. https://doi.org/10.1248/yakushi1947.120.10_899 DOI: https://doi.org/10.1248/yakushi1947.120.10_899
Hou, G.-Q.; Liang, X.-L.; Chen, R.; Tang, L.; Wang, Y.; Xu, P.-Y.; Zhang, Y.-R.; Ou, C.-H. Copper transportion of WD protein in hepatocytes from Wilson disease patients in vitro. World, J. Gastroenterol. 2001, 7, 846–851.
https://doi.org/10.3748/wjg.v7.i6.846 DOI: https://doi.org/10.3748/wjg.v7.i6.846
Siordia-Reyes, A.G.; Ferman-Cano, F.; García, G.R.; Rodríguez-Velasco, A. Wilson disease. Report of a case of autopsy with copper tissue quantification and electronic microscopy. Rev. Gastroenterol Mex 2001, 66, 38–41. PMID: 11464628.
http://www.revistagastroenterologiamexico.org/es-pdf-X0375090601253030
Davie, C.; Schapira, A. Wilson disease. Int. Rev. Neurobiol. 2002, 53, 175–190. https://doi.org/10.1016/S0074-7742(02)53007-5 DOI: https://doi.org/10.1016/S0074-7742(02)53007-5
Shimizu, N. Wilson disease. Nihon Rinsho 2002, 60 (Suppl. 4), 433–436. (In Japanese). PMID: 12013905.
Page, R.A.; Davie, C.A.; MacManus, D.; Miszkiel, K.A.; Walshe, J.M.; Miller, D.H.; Lees, A.J.; Schapira, A. Clinical correlation of brain MRI and MRS abnormalities in patients with Wilson disease. Neurology 2004, 63, 638–643.
https://doi.org/10.1212/01.WNL.0000134793.50831.C1 DOI: https://doi.org/10.1212/01.WNL.0000134793.50831.C1
Roberts, E.A.; Robinson, B.H.; Yang, S. Mitochondrial structure and function in the untreated Jackson toxic milk (tx-j) mouse, a model for Wilson disease. Mol. Genet. Metab. 2008, 93, 54–65.
https://doi.org/10.1016/j.ymgme.2007.08.127 DOI: https://doi.org/10.1016/j.ymgme.2007.08.127
Lee, B.H.; Kim, J.-M.; Heo, S.H.; Mun, J.H.; Kim, J.; Kim, J.H.; Jin, H.Y.; Kim, G.-H.; Choi, J.-H.; Yoo, H.-W. Proteomic analysis of the hepatic tissue of Long-Evans Cinnamon (LEC) rats according to the natural course of Wilson disease. Proteomics 2011, 11, 3698–3705. https://doi.org/10.1002/pmic.201100122 DOI: https://doi.org/10.1002/pmic.201100122
Sauer, S.W.; Merle, U.; Opp, S.; Haas, D.; Hoffmann, G.F.; Stremmel, W.; Okun, J.G. Severe dysfunction of respiratory chain and cholesterol metabolism in Atp7b−/− mice as a model for Wilson disease. Biochim. et Biophys. Acta-Mol. Basis Dis. 2011, 1812, 1607–1615.
https://doi.org/10.1016/j.bbadis.2011.08.011 DOI: https://doi.org/10.1016/j.bbadis.2011.08.011
Zischka, H.; Lichtmannegger, J.; Schmitt, S.; Jägemann, N.; Schulz, S.; Wartini, D.; Jennen, L.; Rust, C.; Larochette, N.; Galluzzi, L.; et al. Liver mitochondrial membrane crosslinking and destruction in a rat model of Wilson disease. J. Clin. Investig. 2011, 121, 1508–1518.
https://doi.org/10.1172/JCI45401 DOI: https://doi.org/10.1172/JCI45401
Lichtmannegger, J.; Leitzinger, C.; Wimmer, R.; Schmitt, S.; Schulz, S.; Kabiri, Y.; Eberhagen, C.; Rieder, T.; Janik, D.; Neff, F.; et al. Methanobactin reverses acute liver failure in a rat model of Wilson disease. J. Clin. Investig. 2016, 126, 2721–2735.
https://doi.org/10.1172/JCI85226 DOI: https://doi.org/10.1172/JCI85226
Zischka, H.; Einer, C. Mitochondrial copper homeostasis and its derailment in Wilson disease. Int. J. Biochem. Cell Biol. 2018, 102, 71–75. https://doi.org/10.1016/j.biocel.2018.07.001 DOI: https://doi.org/10.1016/j.biocel.2018.07.001
Einer, C.; Leitzinger, C.; Lichtmannegger, J.; Eberhagen, C.; Rieder, T.; Borchard, S.; Wimmer, R.; Denk, G.; Popper, B.; Neff, F.; et al. A high-calorie diet aggravates mitochondrial dysfunction and triggers severe liver damage in Wilson disease rats. Cell. Mol. Gastroenterol. Hepatol. 2018, 7, 571–596.
https://doi.org/10.1016/j.jcmgh.2018.12.005 DOI: https://doi.org/10.1016/j.jcmgh.2018.12.005
Polishchuk, E.V.; Merolla, A.; Lichtmannegger, J.; Romano, A.; Indrieri, A.; Ilyechova, E.Y.; Concilli, M.; De Cegli, R.; Crispino, R.; Mariniello, M.; et al. Activation of autophagy observed in liver tissues from patients with Wilson disease and from ATP7B-deficient animals, protects hepatocytes from copper-induced apoptosis. Gastroenterology 2019, 156, 1173–1189.e5.
https://doi.org/10.1053/j.gastro.2018.11.032 DOI: https://doi.org/10.1053/j.gastro.2018.11.032
To, U.; Schilsky, M.L. A Case for Not Going Global: “Americanization” of diet accelerates hepatic mitochondrial injury in a model of Wilson disease. Cell. Mol. Gastroenterol. Hepatol. 2019, 7, 684–685. https://doi.org/10.1016/j.jcmgh.2019.01.001 DOI: https://doi.org/10.1016/j.jcmgh.2019.01.001
Zhang, J.; Tang, L.-L.; Li, L.-Y.; Cui, S.-W.; Jin, S.; Chen, H.-Z.; Yang, W.-M.; Xie, D.-J.; Yu, G.-R. Gandouling tablets inhibit excessive mitophagy in toxic milk (TX) model mouse of Wilson disease via Pink1/Parkin pathway. Evidence-Based Complement. Altern. Med. 2020, 2020, 1–11.
https://doi.org/10.1155/2020/3183714 DOI: https://doi.org/10.1155/2020/3183714
Agarwal, M.; Saba, L.; Gupta, S.K.; Johri, A.M.; Khanna, N.N.; Mavrogeni, S.; Laird, J.R.; Pareek, G.; Miner, M.; Sfikakis, P.P.; et al. Wilson disease tissue classification and characterization using seven artificial intelligence models embedded with 3D optimization paradigm on a weak training brain magnetic resonance imaging datasets: A supercomputer application. Med. Biol. Eng. Comput. 2021, 59, 511–533. https://doi.org/10.1007/s11517-021-02322-0 DOI: https://doi.org/10.1007/s11517-021-02322-0
Record, C.O.; Buxton, B.; Chase, R.A.; Curzon, G.; Murray-Lyon, I.M.; Williams, R. Plasma and brain amino acids in fulminant hepatic failure and their relationship to hepatic encephalopathy. Eur J. Clin. Investig. 1976, 6, 387–394.
https://doi.org/10.1111/j.1365-2362.1976.tb00533.x DOI: https://doi.org/10.1111/j.1365-2362.1976.tb00533.x
Fabbri, A.; Magrini, N.; Bianchi, G.; Zoli, M.; Marchesini, G. Overview of randomized clinical trials of oral branched-Chain amino acid treatment in chronic hepatic encephalopathy. J. Parenter. Enter. Nutr. 1996, 20, 159–164.
https://doi.org/10.1177/0148607196020002159 DOI: https://doi.org/10.1177/0148607196020002159
Reilly, J.; Mehta, R.; Teperman, L.; Cemaj, S.; Tzakis, A.; Yanaga, K.; Ritter, P.; Rezak, A.; Makowka, L. Nutritional support after liver transplantation: A randomized prospective study. J. Parenter. Enter. Nutr. 1990, 14, 386–391.
https://doi.org/10.1177/0148607190014004386 DOI: https://doi.org/10.1177/0148607190014004386
Amodio, P.; Canesso, F.; Montagnese, S. Dietary management of hepatic encephalopathy revisited. Curr. Opin. Clin. Nutr. Metab. Care 2014, 17, 448–452.
https://doi.org/10.1097/MCO.0000000000000084 DOI: https://doi.org/10.1097/MCO.0000000000000084
Gluud, L.L.; Dam, G.; Borre, M.; Les, I.; Cordoba, J.; Marchesini, G.; Aagaard, N.K.; Risum, N.; Vilstrup, H. Oral branched-chain amino acids have a beneficial effect on manifestations of hepatic encephalopathy in a systematic review with meta-analyses of randomized controlled trials. J. Nutr. 2013, 143, 1263–1268. https://doi.org/10.3945/jn.113.174375 DOI: https://doi.org/10.3945/jn.113.174375
Park, J.G.; Tak, W.Y.; Park, S.Y.; Kweon, Y.O.; Chung, W.J.; Jang, B.K.; Bae, S.H.; Lee, H.J.; Jang, J.Y.; Suk, K.T.; et al. Effects of Branched-Chain Amino Acid (BCAA) Supplementation on the progression of advanced liver disease: A Korean nationwide, multicenter, prospective, observational, cohort study. Nutrients 2020, 12, 1429. https://doi.org/10.3390/nu12051429 DOI: https://doi.org/10.3390/nu12051429
Vidot, H.; Cvejic, E.; Finegan, L.J.; Shores, E.A.; Bowen, D.G.; Strasser, S.I.; McCaughan, G.W.; Carey, S.; Allman-Farinelli, M.; Shackel, N.A. Supplementation with synbiotics and/or branched chain amino acids in hepatic encephalopathy: A pilot randomised placebo-controlled clinical study. Nutrients 2019, 11, 1810.
https://doi.org/10.3390/nu11081810 DOI: https://doi.org/10.3390/nu11081810
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2023 Medici, V.; Czlonkowska, A.; Litwin, T.; Giulivi, C.

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-SinDerivadas 4.0.
Magna Scientia UCEVA proporciona un acceso abierto, libre y gratuito a su contenido, basado en el principio de que ofrecer al público un acceso libre a las investigaciones, ayuda a un mayor intercambio global del conocimiento. Lo cual, implica que los usuarios pueden leer, descargar, almacenar, imprimir, buscar, indexar y realizar enlaces a los textos completos de esta revista. Se permite distribuir los diversos artículos en las versiones post-print y oficial, sin previo permiso del autor o editor, considerando que el fin de este, no implica fines comerciales, ni la generación de obras derivadas; Solo se solicita la mención de la fuente así como la autoría. El titular del copyright será el o los autores que publiquen en Magna Scientia UCEVA.
Magna Scientia UCEVA está distribuida bajo los términos de la licencia https://creativecommons.org/licenses/by-nc-nd/4.0/deed.es